8.3 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS EUCARIÓTICOS.
Los tipos de señales que pueden alterar la
transcripción de un gen puede ser:
Las señales
nutricionales e iónicas suelen darse en eucariotas unicelulares solamente
porque son los únicos a los que va a afectar el medio en el que están
creciendo. La excepción se encuentra en los hepatocitos (es el regulador de la
concentración sanguínea de muchos metabolitos) y las células en cultivo.
Señales hormonales que
interaccionan con un receptor de la membrana. En la mayoría de los casos, la
señal externa provoca la aparición del segundo mensajero intracelular. La
cascada de transducción de señal subsiguiente produce un regulador de
transcripción específico.
En el caso de las hormonas esteroideas, el receptor está dentro de la célula y
es el conjunto hormona-receptor el que actúa de regulador.
Proteínas que modulan la transcripción
En eucariotas,
pueden actuar como reguladores tanto moléculas de RNA como proteínas. Entre las
proteínas, las hay que forman parte de la holoenzima polimerasa (los factores
de transcripción), otras intervienen en la remodelación de la cromatina y un
tercer grupo se une al DNA para regular la transcripción, que es el que nos
ocupa.
Los mecanismos que regulan el proceso de expresión de la información
genética operan a varios niveles. Un primer nivel de regulación
viene dado por el acceso al ADN. La cromatina se presenta en dos formas, una
muy condensada llamada heterocromatina y una más laxa llamada eucromatina,
siendo esta última la que representa la fracción activa del ADN.
Mientras más compactada está la cromatina más inaccesible
resulta el ADN a las proteínas que deben realizar la transcripción.
1.-
Activadores transcripcionales
Los activadores son la proteínas que se van a
unir a los elementos distales (SDE y potenciadores) para activar la
transcripción. Son específicos de unos pocos promotores —por lo que no
estarán en todos los tipos celulares—, reconocen entre 6 y 14 pb en el
promotor y suelen tener dos dominios estructurales:
- El dominio de unión a DNA (DNA binding
domain) , que consta de 60 a 100 aminoácidos consecutivos.
- El dominio de activación de la transcripción
que consta de 30 a 100 aminoácidos que no tienen por qué ser consecutivos.
- La presencia de estos dominios las convierte en proteínas
modulares en las que el dominio de unión y el de activación pueden
funcionar independientemente.
2.-
Coactivadores y correpresores
Se denomina correpresor si ayuda
a inactivar el promotor. Los correpresores pueden tener actividad desacetilasa,
con lo que hace que el DNA se una con más firmeza a los nucleosomas,
inactivando el promotor porque no puede ser reconocido por los factores
generales de transcripción. Entre los más conocidos podemos encontrar SMRT
(correpresor del receptor de hormonas tiroideas) y N-Cor (correpresor del
receptor hormonal nuclear), formados por un único péptido.
Se denomina coactivador si ayuda
a activar la transcripción. Un mismo coactivador puede recibir señales de
distintos activadores para transmitirlos hacia el complejo del promotor basal.
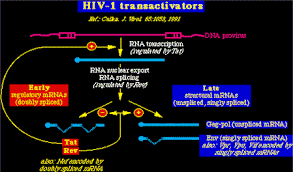
3.- Transactivadores
Un transactivador es un elemento proteico capaz de activar la
transcripcion y por lo tanto la traduccion y expresion de determinada
proteina. Los virus pueden usar estos elementos para desregular la
expresion proteica y asegurarse la provision de ciertas proteinas
necesarias para realizar su ciclo infectivo.
Son aquellos que directamente ejercen su acción
interaccionando con el complejo de iniciación formado en el promtor basal, bien
sobre la propia polimerasa o, más normalmente, sobre una de las TAF o de los
TFII, para activar o reprimir la transcripción, puesto que no son actividades
exclulyentes.
Potenciadores
Potenciadores de ADN regulador función de los elementos que más de
distancia variable de alterar el nivel de la expresión génica. Un
potenciador generalmente no es suficiente para la expresión de genes y
no requiere promotor elementos para iniciar la transcripción. Enhancer
elementos proporcionar gran parte del espacio, el tiempo y el medio
ambiente regulada la expresión de genes de plantas.

Silenciamiento de genes
La unión inespecífica de proteínas reguladoras es
un problema importante en los organismos con genomas grandes. Para combatirla,
los eucariotas han hecho que los genes tengan en torno a 5 dianas para
proteínas reguladoras diferentes. Esta estrategia es útil para los activadores
de la transcripción porque es una estrategia eficiente y ahorra esfuerzo. Una
estrategia similar no es factible con los inhibidores de la transcripción, por
lo que se da poca regulación por silenciamiento.
Un gen se transcribe a un ARN mensajero (ARNm) en el núcleo o lo que es
lo mismo se expresa, tras ser procesado sale del núcleo y una vez en el
citoplasma es traducido a proteína por los ribosomas. Las proteínas son
las que en última instancia realizan el trabajo molecular y por tanto
son las dianas de nuestros fármacos. Por ello buscamos sustancias que
bloqueen o cambien su actividad, para obtener efectos terapéuticos. Pero
que pasaría si cambiáramos de objetivo y pudiéramos actuar a nivel de
la traducción, ese es el enfoque de los medicamentos de ARN.
El silenciamiento de un gen puede ocurrir por:
- la inactivación por interacción con un regulador
- el silenciamiento génico postranscripcional (PTGS, también
denominada cosupresión o extinción génica)
- la metilación del DNA en vertebrados (directamente ligada al
superenrollamiento y al silenciamiento).
Inactivación
mediante una proteína reguladora
Se consigue uniendo una proteína reguladora a
cualquiera de los distintos elementos que forman los promotores.
Los que reconocen
los elementos distales
• el silenciador
específico de tejido (tse): se encarga de silenciar en cualquier célula
los genes que son específicos de células hepáticas
• las hormonas esteroideas comentadas anteriormente
• el gen Pit-1
Los que reconocen
los elementos proximales
• la proteína CDPC: recibe el nombre de «desplazamiento de
CAAT» porque impide que la caja CAAT sea reconocida por sus proteínas
específicas
Los que reconocen
el promotor basal
• el represor global Dr1/DRAP1: es un heterodímero que se une
a TBP para evitar que interactúe con TFIIB
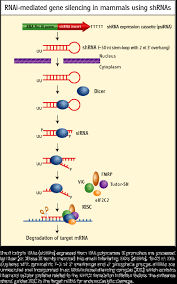
PTGS:
silenciamiento génico postranscripcional
Consiste en la degradación específica de los mRNA complementarios de
una de las hebras del dsRNA. Los mRNA degradados suelen ser
transcritos aberrantes de diversos orígenes. También se denomina cosupresión
o extinción (quelling). Este RNA aberrante es sustrato de una
RNA-polimerasa dirigida por RNA que genera una larga molécula de dsRNA, conocida
con el nombre de dsRNA desencadenante. Éste es fragmentado por la
ribonucleasa Dícer
en una serie de dsRNA de 21 a 25 nucleótidos de longitud denominados «RNA
interferentes pequeños» (siRNA). Este siRNA se asocia a una serie de
proteínas para formar el complejo RISC (RNA-induced silencing
complex). En este complejo, una de las hebras del siRNA sirve de guía para
localizar cualquier mRNA complementario presente en la célula con vistas a su
destrucción mediante una endorribonucleasa del complejo RISC (tentativamente
llamada slicer).
Se trata de un mecanismo extremadamente
conservado entre los organismos eucariotas (protozoarios, mamíferos, plantas,
peces, insectos, hongos, invertebrados y seres humanos) por lo que puede tratarse
de un mecanismo de regulación y defensa que tuvo su origen en el mundo RNA.
Desempeña un papel fundamental en varios procesos celulares:
- Defensa contra la invasión de ácidos nucleicos intrusos
(normalmente virus)
- Integridad del genoma, y aque reprime la transposición de los
elementos móviles
- Destrucción de mRNA aberrantes que generarían desconcierto
intracelular
- Mantenimiento de las zonas superenrolladas (heterocromatina)
del genoma (véase más adelante en el silenciamiento por metilación).
Mientras en algunos organismos (por ejemplo, en
las células humanas) se manifiesta como un fenómeno transitorio (que cede con
la desaparición del dsRNA exógeno desencadenante), en otros (plantas y
nematodos), se amplifica y difunde hacia el resto de las células del organismo,
pudiendo llegar a ser heredable, al menos por algunas generaciones (en Drosophila
y en nematodos, pero no en plantas).

BIBLIOGRAFIA

